A UNIX típusú rendszerekben a fork() a mitózis.
Az témával kapcsolatos feladatok "széles" spektrumával tervezünk gyakorlatozni a laborokon, ez megér egy külön posztot, hogy ne csak a trófeák közti vadászatban bogarásszuk őket :)
Rántsuk le at emberi genom 2. kromoszómájának egy részét: ftp.ncbi.nlm.nih.gov/genomes/H_sapiens/Assembled_chromosomes/seq/
T, C, A, G betűkkel találkozunk, ahogyan hármasával egy fehérjét kódolnak. A következő kis progival (mivel nem feledhetjük, hogy kezdő programozók is vannak köztünk, ezért az ilyen egyszerűekkel kezdünk) tehát a következő kis progival átkódoljuk a sorozatot, a Javát tanítok www.tankonyvtar.hu/informatika/javat-tanitok-1-1-1-080904 TCAG2Hexa osztályának mintájára két nukleotid bázisból egy hexadecimális betűt készítünk:
#include <stdio.h> #include <unistd.h> int main (void) { char hexa_jegyek[] = {'A', 'B', 'C', 'D', 'E', 'F'}; int paratlan = 0, elso=0, masodik=0, i = 0; while((i=getchar()) != EOF) { switch(i) { case 'T': masodik = 0; break; case 'C': masodik = 1; break; case 'A': masodik = 2; break; case 'G': masodik = 3; break; } if(paratlan) { int jegy = 4*elso + masodik; if(jegy<10) printf("%d", jegy); else printf("%c", hexa_jegyek[jegy-10]); } paratlan = !paratlan; elso = masodik; } }
Az alábbi kis játék során megismerkedünk a (2. előadásban bevezetett) CompLearn csomag használatával.
- Rántsuk le a humán genom 2. kromoszómájának első 1400 betűjét ftp.ncbi.nlm.nih.gov/genomes/H_sapiens/CHR_02/ (amit a hs_alt_Hs_Celera_chr2.fa.gz név alapján Hschr21400 néven mentünk most el, alul majd helso.human1minta)!
- Generáljunk le 1000 hexa jegyet a Pi kifejtésének 1.000.000. jegyétől (piezer.txt, alul majd piezer.1000000)!
- Az első előadás pszeudorandom kártyájával és a fenti tcag2hex kiírás további finomításával
#include <stdio.h> #include <stdlib.h> #include <time.h> #include <sys/types.h> #include <unistd.h> int main (void) { int i, r; srand (time (NULL) + getpid ()); for (i = 0; i < 2000; ++i) { r = (int) (16.0 * rand () / (RAND_MAX + 1.0)); if (r < 10) printf ("%d", r); else printf ("%c", r - 10 + 'A'); } printf ("\n"); return 0; }
nyomtassunk ki 2000 db véletlen jegyet (veletlen2000, alul majd rand.2000db)! - Rántsunk még le a 3. emberi koromóból is 700 betűt (Hschr3700)!
- A C elegans fonalféreg II. kromójából is vegyünk valamekkora mintát (c2_2000) ftp.ncbi.nlm.nih.gov/genomes/Caenorhabditis_elegans/CHR_II/NC_003280.fna (ez a féreg közel áll a szívünkhöz az Orch OR tudatmodell miatt is, alul majd celeg.celegans)
- ... s készítünk még pár mintát... a kutya (canis familiaris), a ló (equus caballus), a kakas (gallus gallus), a szarvasmarha (bos taurus) 2. kromójának elejéből, a Pi kifejtéséből, a csupa 1 jegyből, a 01-ekből álló állományokból:
batfai@kalapacs:~ $ ls -l mintak/S lássuk, mit mutat ezekre a mintákra a CompLearn normalizált tömörítési távolsága alapján készített gráfja:
összesen 72
-rw-r--r-- 1 batfai batfai 2100 2011-02-27 18:49 celeg.celegans
-rw-r--r-- 1 batfai batfai 800 2011-02-27 18:20 egyes.8001es
-rw-r--r-- 1 batfai batfai 3220 2011-02-27 21:14 h11kromo.eleje
-rw-r--r-- 1 batfai batfai 3080 2011-02-27 21:17 h11kromo.kozepe
-rw-r--r-- 1 batfai batfai 3260 2011-02-27 21:16 h11kromo.vege
-rw-r--r-- 1 batfai batfai 1400 2011-02-27 17:24 helso.human1minta
-rw-r--r-- 1 batfai batfai 700 2011-02-27 17:24 hmaso.human2minta
-rw-r--r-- 1 batfai batfai 3448 2011-02-27 18:44 kakas.2k
-rw-r--r-- 1 batfai batfai 1400 2011-02-27 21:02 kutya.1400
-rw-r--r-- 1 batfai batfai 1190 2011-02-27 21:09 kutya.kozepe
-rw-r--r-- 1 batfai batfai 1127 2011-02-27 21:09 kutya.vege
-rw-r--r-- 1 batfai batfai 1190 2011-02-27 18:45 lo.2k
-rw-r--r-- 1 batfai batfai 1600 2011-02-27 18:27 nulegy.800par
-rw-r--r-- 1 batfai batfai 1000 2011-02-27 18:39 pi1000.0tol
-rw-r--r-- 1 batfai batfai 2000 2011-02-27 18:35 pi2000.0tol
-rw-r--r-- 1 batfai batfai 1000 2011-02-27 17:25 piezer.1000000
-rw-r--r-- 1 batfai batfai 2000 2011-02-27 17:24 rand.2000db
-rw-r--r-- 1 batfai batfai 700 2011-02-27 20:57 szarvasm.700
batfai@kalapacs:~ $
ncd -b -d mintak/ mintak/
maketree distmatrix.clb
neato -Tpng treefile.dot > tcag.png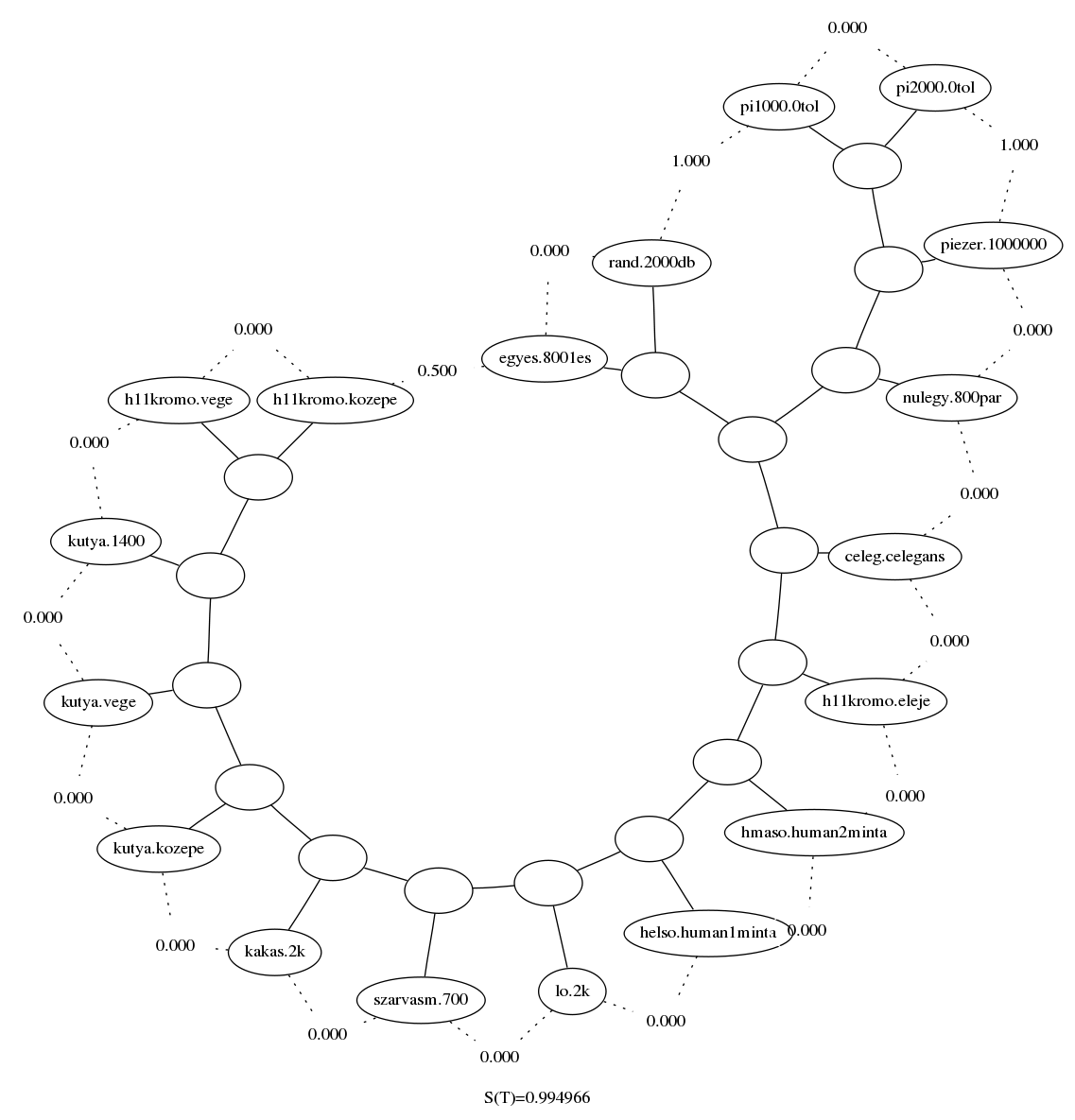
Teker a tovább linkre, ha kíváncsi vagy a folytatásra!
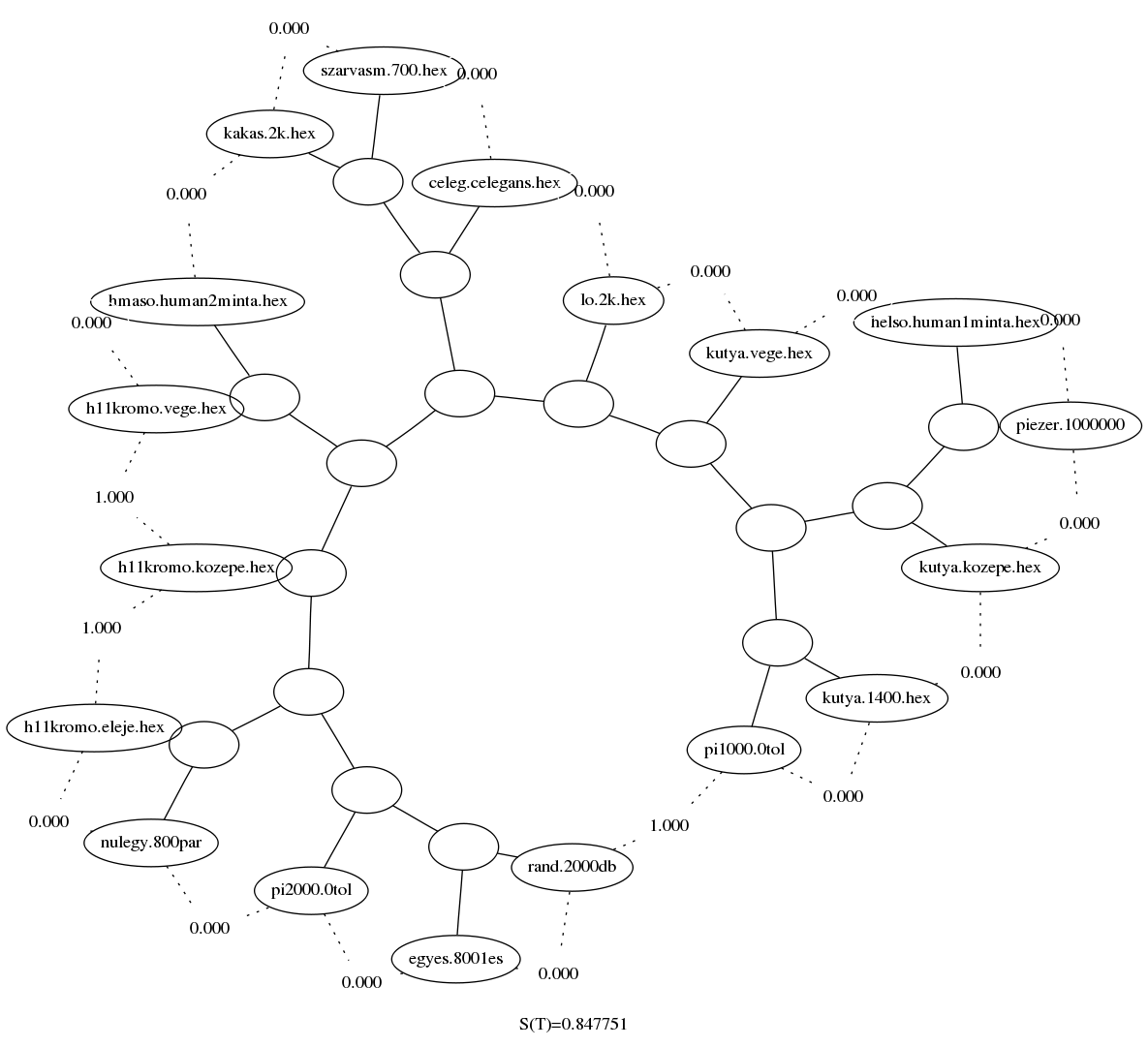
A poszt elején említett kódolást a lerántott genetikai kódrészletekre alkalmazva kapjuk a .hex állományokat, ezzel így módosul a gráf:
Bajnokság van, nevezel?
Néhány a genetikai kóddal kapcsolatos kisbajnokság már eddig is szerepelt a trófeák között, de íme néhány új:
- 2 trófea annak, aki először megmondja, hogy mennyi az LZW fa ághosszainak szórása a humán 2 kromóban lévő genetikai kódnak. A kódot bináris állományként dolgozd fel (előtte persze azért tömörítsd ki :), jöhet kommentben is a megoldás. (A szórást a Jávácska ONE-beli Hetedik Szem (javacska-one-1.0.0-projects.zip / hetedik-szem-1.0.0 / src / main / java / TudatSzamitas.java) kódja alapján fejleszd bele a korábbi kódunkba.)
- 3 trófea annak, aki először megmondja, hogy mennyi az LZW fa ághosszainak szórása a humán 2 kromóban lévő genetikai kódnak. A kódot tedd át 2 betűnként hexába, aztán a hexa betűkből nyomj 4 bitet betűnként, az így kapott állományt vizsgáld, jöhet kommentben is a megoldás. (A szórást a Jávácska ONE-beli Hetedik Szem (javacska-one-1.0.0-projects.zip / hetedik-szem-1.0.0 / src / main / java / TudatSzamitas.java) kódja alapján fejleszd bele a korábbi kódunkba.)
- 4 trófea annak, aki először megmondja, hogy mennyi az LZW fa ághosszainak szórása a humán 2 kromóban lévő genetikai kódnak. A kódot karakteresen a szereplő 5 betű alapján vizsgáld, jöhet kommentben is a megoldás. (A szórást a Jávácska ONE-beli Hetedik Szem (javacska-one-1.0.0-projects.zip / hetedik-szem-1.0.0 / src / main / java / TudatSzamitas.java) kódja alapján fejleszd bele a korábbi kódunkba. A korábbi LZW-s kódunkba ne bináris fát építs, hanem olyat, aminek egy csomópontjából 5 mutató mutat a lehetséges 5 betűvel címkézett gyerek csomópontokra.)
- (s egy kicsit más jellegű feladat) 1 trófea annak, aki a genetikai kódot a kódnak megfelelő fehérjék sorozatává kódolja, pl.: TGT=Cys, TGG=Trp, TGA=STOP, segít a DNA codon table, jöhet kommentben a kód és egy kis rövid demó minta, hogy lássam jó-e.